Adv Sci:新的深度学习模型有望彻底改变转录组学中的CircRNA分析
来源:
|
作者:李
|
发布时间: 705天前
|
259 次浏览
|
分享到:

环状RNA(circRNAs)长期以来被认为是一类至关重要但相对未被探索的转录本,以其组织和细胞类型特异性表达模式而闻名。然而,尽管单细胞和空间转录组学取得了进步,但circRNA测序效率的固有限制阻碍了circRNA的有效分析。为了解决这一研究中的关键空白,研究人员提出了一种深度学习模型CIRI-deep,用于全面预测各种类型的RNA-seq数据上的circRNA调控。相关研究成果在杂志Advanced Science上发表,题为“CIRI-Deep Enables Single-Cell and Spatial Transcriptomic Analysis of Circular RNAs with Deep Learning”
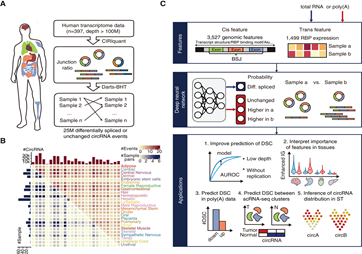
图源:CIRI-Deep Enables Single-Cell and Spatial Transcriptomic Analysis of Circular
RNAs with Deep Learning ,DOI: 10.1002/advs.202308115
CIRI-deep代表了circRNA分析的重大飞跃。在包含2500万个高置信度circRNA调控事件的广泛数据集上进行训练,该模型在测试和遗漏数据上都表现出出色的性能,确保了从RNA-seq数据集推断差异事件的无与伦比的准确性。CIRI-deep的多功能性超越了传统的circRNA分析方法。该模型及其改编版本能够进行广泛的circRNA分析,包括集群或区域特异性circRNA检测、后剪接结(BSJ)比率图的可视化以及trans和cis特征重要性的评估。此外,它的适应性涵盖了所有主要类型的RNA-seq数据集,包括单细胞和空间转录组数据,从而为circRNA研究开辟了新的途径。
本研究将CIRI-Deep描述为在高维转录组数据集中研究circRNA的强大工具,使研究人员能够揭示circRNA在不同生物学背景下的调控作用和表达模式。深度学习技术与circRNA分析的结合为促进我们对circRNA生物学及其对健康和疾病的影响的理解更近一步。
声明:本网所有转载文章内容为了宣传行业动态所用,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。